M1 MABS Graphes TP Recherche de communautés dans les graphes
From silico.biotoul.fr
Contexte biologique
La grande disponibilité de données issues de séquençage de génomes complets rend les approches basées sur la génomique comparative particulièrement attractives pour aborder les questions relatives à l’adaptation des bactéries à leur environnement. Dans ce cadre, les systèmes codés par de grandes familles de gènes paralogues, comme les transporteurs ABC, sont ceux qui présentent le plus grand potentiel. En effet, ces systèmes sont impliqués dans l'import ou l'export de molécules diverses à travers la membrane cytoplasmique. Ils sont ubiquitaires chez les archaea et les bactéries, ils seraient donc apparus très tôt dans l’évolution. Cependant, ils peuvent avoir des distributions différentes même entre génomes apparentés suggérant des vagues successives de gains/pertes de gènes.
Les transporteurs ABC peuvent être classés en une trentaine de sous-familles aux quelles nous pouvons associer des types de substrats de nature spécifiques (sucres, acides aminées, peptides, ions, ferrichromes...). Cependant, nous disposons de très peu d'informations sur la nature exacte de la molécule transportée par un transporteur. Or cette information est nécessaire si l'on veut pouvoir étudier la contribution de ces transporteurs à l'adaptation des bactéries à leur environnement. Une approche bioinformatique pour tenter de répondre à cette question consiste à regrouper les différents systèmes en groupe de gènes orthologues. Sous l'hypothèse que ces gènes orthologues ont conservé la même fonction, et si le substrat transporté par le produit d'un des gènes d'un groupe a été identifié expérimentalement, alors on peut propager cette annotation à tous les autres membres du groupe. Une autre approche consiste a exploiter le contexte génomique. En effet, une forte association entre les gènes appartenant au même groupe d'orthologues et un autre gène peut révéler un lien fonctionnel entre le transporteur et le produit de ce gène, comme par exemple, si le gène code pour une enzyme, alors on peut penser que le substrat de cette enzyme a de forte chance d'être la molécule prise en charge par le transporteur ABC.
Cette approche nécessite comme préalable l'identification de groupes de gènes orthologues. Si cela peut paraitre assez facile à réaliser dans le cas de gènes présentant pas ou peu de duplication dans les génomes, la tache est beaucoup plus difficile à effectuer avec des familles de gènes fortement paralogues. Néanmoins, dans tous les cas, il est nécessaire d’établir de façon précise les liens de parenté entre les séquences. Cela peu se faire à l’aide de représentation arborée ou à l’aide de graphes. Les arbres fournissent une bonne modélisation des trajectoires évolutives des séquences mais sont très sensibles à la qualité des données utilisées pour les construire. A l’inverse, les graphes sont plus difficiles à manipuler et à interpréter, mais présentent l’avantage d’être beaucoup plus robustes et de pouvoir inclure un très grand nombre de données. Généralement, les gènes (ou les protéines) constituent les sommets du graphe. Ils sont reliés par des arcs pouvant traduire une relation évolutive entre les gènes. Ces liens sont qualitatifs (homologie, orthologie, paralogie). Si les données sont suffisamment dissemblables, le graphe peut se décomposer en un ensemble de composantes connexes (ensemble de sommets connectés par au moins un arc), on a alors une classification de type COG. Sinon, les groupes de gènes orthologues ne sont pas déconnectés dans le graphe, mais correspondent à des régions de plus forte densité (communautés). L’identification de groupes de gènes orthologues revient alors à l’identification de communautés dans un graphe.
Jeu de données
A titre d’exemple, nous allons analyser une sous familles de transporteurs ABC, sous famille 1, dont les membres sont impliqués dans l’import de sucre. A l’aide de la base de données ABCdb, nous avons sélectionné une liste de transporteurs de la sous famille 1. Pour chaque transporteur, nous avons extrait les protéines codées par les gènes, localisés sur le chromosome, en amont et en aval des gènes codant pour la transporteur (4 gènes de par et d’autre). Pour toutes ces protéines, nous disposons des pairs de liens d’isorthologie.
Remarques:
- Les liens d’homologies n’ont de sens qu’au niveau des gènes, mais ils ont été établis au niveau des protéines en raison de leur meilleure conservation.
- Les gènes orthologues a et b, appartenant aux génomes A et B, sont isorthologues s'il n'existe pas de paires de gènes paralogues (a, y) dans A et (x, b) dans B avec une distance phylogénétique inférieure à celle de la paire (a, b). La liste de ces pairs d’isorthologues constitue la liste d’arête d’un graphe dont les sommets sont les protéines.
- Dans la base de données ABCdb, les noms des protéines sont construits de la façon suivante:
- Première lettre en majuscule : la première lettre du genre (E pour Escherichia)
- Les trois lettres suivantes en minuscule : les trois premières lettres de l’espèce (col pour coli)
- La cinquième lettre majuscule est le code souche (A pour K12)
- Les deux chiffres suivant se réfèrent au numéro du chromosome (01 pour le chromosome 1)
- Un point est utilisé comme séparateur, il est suivit du nom de la protéine
Exemple : EcolA01.RBSA
- Les annotations des gènes/protéines sont compilées dans un fichier, dont les colonnes correspondent à :
- le nom de la protéine
- annotation COG
- le non du gène (protéine) d'ancrage, utilisé comme référence pour la région chromosomique contenant les gènes codant pour un transporteur ABC,
- le nom du système ABC (le nom d'un des partenaires),
- le domaine (NBD, MSD, SBP)
- sous famille
Fichiers
Mise en œuvre
Nous allons utiliser la librairie : iGraph et la librairie ade4.
Prise en main du graphe
La fonction read.graph permet de lire la liste des arêtes à partir d’un fichier.
graph_file <- 'http://silico.biotoul.fr/site/images/0/0b/Cleandb_Luca_1_A_1_1_65_Iso_Tr_1.gr' graph <- read.graph(graph_file, format = "ncol", directed = FALSE)
Nous allons utiliser les fonctions vcount et ecount pour compter le nombre de sommets et d’arêtes du graphe.
Avant de poursuivre, il est nécessaire d’appliquer la fonction simplify() sur notre graphe.
*Quelle est l’effet de cette fonction ? *Discutez cette observation en la ramenant à la façon dont a été construit le graphe.
Dans la suite de l’exercice nous utiliserons le graphe 'simplifié'.
Taux de paralogie
On s’attend à ce qu’un assemblage de gènes orthologues 1:1 ne comporte pas de gènes paralogues. Nous allons donc écrire une fonction qui calcule le taux de gènes paralogues dans une liste de gènes. Pour cela nous allons compter le nombre de gènes présent dans la liste et le comparer au nombre de souches. S’il n’y a pas de paralogue, chaque souche est représentée par un seul gènes, sinon, on peut avoir plus d'un gène par souche. Ainsi, pour obtenir le taux de paralogues, on divise la différence entre nombre de gènes et nombre de souches par le nombre de gènes.
Les fonctions à utiliser sont : substr() et unique(). L’expression V(graph)$name permet d’obtenir la liste des sommets du graphe.
count_paralogs <-function(list) { ... return(paralogs) }
Utilisez cette fonction pour calculer le taux de paralogues dans le graphe.
*Quelle peut être l’origine de ce taux élevé de paralogie ?
Représentation graphique
Nous allons observer graphiquement le graphe. Pour faciliter les comparaisons entre différentes « colorations » du graphe, nous allons fixer la disposition (le layout) du graphe à l’aide de l’expression suivante :
globalGraphLayout = layout.fruchterman.reingold(graph) plot(graph, layout=globalGraphLayout, vertex.label=NA, vertex.size=3)
*Décrire le graphe obtenu.
Annotations
Nous allons utiliser les annotations en domaines contenu dans le fichier annot_file pour colorier le graphe.
Lecture du fichier :
annot_file <- 'http://silico.biotoul.fr/site/images/1/19/Annotation.txt' annotation <- read.table(annot_file, header=TRUE, row.names=1)
Annotation en domaines
Les colonnes du fichier sont : 'protein name', 'anchor', 'cog', 'assembly', 'domain', 'subfamily'. La difficulté est que certaines protéines n’ont pas de domaines annotés (ils ne sont pas partenaire d'un système ABC) dans le fichier.
library(knitr) kable(head(annotation))
domains = annotation[V(graph)$name, 4]
Pensez à associer une couleur à chaque domaine (color = rainbow(x)).
plot(graph, layout=globalGraphLayout, vertex.color=domains, vertex.label=NA, vertex.size=3) legend("topright", legend=levels(domains), cex=0.7, fill=color)
Annotation en sous familles
ssfam = annotation[V(graph)$name, 5];
* Commentez les résultats obtenus.
Extraction des composantes connexes
Nous allons maintenant extraire les composantes connexes (CC) du graphe à l’aide de la fonction decompose.graph() en utilisant le mode = "strong".
Nous ne conserverons que les CC contenant au moins 5 sommets. La liste des objets graphe sera conservée dans la variable cc.
* Combien de composantes connexes avez-vous obtenu ?
Propriétés des composantes connexes
Les CC apparaissent hétérogènes au niveau de leur taille mais aussi au niveau de leur densité en arêtes.
* Quelle est le nombre d’arêtes maximum possible dans un graphe de n sommets ?
Vous pouvez établir une fonction donnant le nombre d’arêtes maximum d’un graphe en fonction du nombre de sommets. Nous allons donc étudier la distribution du nombre d’arêtes en fonction du nombre de sommets de nos CC et nous allons tracer la courbe de la fonction d’arêtes maximum.
Distribution du nombre d’arêtes en fonction du nombre de sommets
On peut par exemple placer ces informations dans un tableau :
cc_info <- array(0, c(nb_cc, 2)) for ( i in 1:nb_cc ) { cc_info[i,1] <- vcount(cc[[i]]) cc_info[i,2] <- ecount(cc[[i]]) }
cc[[i]]
Nombre d’arêtes maximum
Puis calculer la courbe de la fonction:
maxx <- max(cc_info[,1]) maxy <- max(cc_info[,2]) courbe <- c() i <- 0 j <- 0 while(j < maxy && i < maxx ) { i <- i + 1 j <- i*(i-1)/2 courbe[i] <- j }
On pourra utiliser la fonction points() pour ajouter la courbe à la distribution des CC.
* Commentez le résultat obtenu.
Taux de paralogies
Pour compléter l’analyse, nous allons ajouter le taux de paralogies de chaque CC.
paralogs <- array(0, c(nb_cc, 2)) for ( i in 1:nb_cc ) { list_name <- V(cc[[i]])$name paralogs[i, 1] <- vcount(cc[[i]]) paralogs[i, 2] <- count_paralogs(list_name) }
Pour superposer le résultat sur le graphique précédant, nous pouvons utiliser :
par(new=T) plot(paralogs, pch=18, col='red', axes=F, xlim=xlim, ylim=zlim, ,xlab= '', ylab='') axis(side=4) mtext('paralogs', side=4, line=2, col='red')
Il peut être nécessaire d’augmenter la place disponible à droite du graphique pour afficher la légende (fonction mar() à utiliser dans un par()).
* Quelle propriété du graphe semble liée à la distribution du taux de paralogie ?
Taux de paralogie et modularité
Nous allons étudier la distribution du taux de paralogie en fonction de la modularité des CC. Pour cela nous allons utiliser fastgreedy.community() qui permet de faire le découpage en communautés d’un graphe.
Remarque: dans les versions antérieures de igraph, il était nécessaire d'utiliser get.adjlist() pour passer des noms des protéines à la liste des sommets.
modularity <- c() for ( i in 1:nb_cc ) { com <- fastgreedy.community(cc[[i]], merges=TRUE, modularity=TRUE) modularity[i] <- max(com$modularity) }
* Que pouvez-vous en conclure?
Relation entre voisinage des gènes et découpage en CC
Nous avons vu que les gènes codant pour différents partenaires de transporteurs ABC étaient très souvent co-localisés sur le chromosome. Dans notre analyse nous avons ajouté 4 gènes en aval et en amont des gènes de chaque système.
Nous allons récupérer les informations des nœuds du graphe à partir d'annotation. La difficulté est que ce data.frame contient beaucoup plus d'informations que celles relatives aux sommets du graphe. Il est donc nécessaire de filtrer annotation pour ne retenir que les lignes associées aux sommets du graphe.
Nous allons conserver ces information dans un data.frame dont les noms de lignes correspondent aux noms des sommets. Les colonnes de 1à 5 sont celles d'annotation.
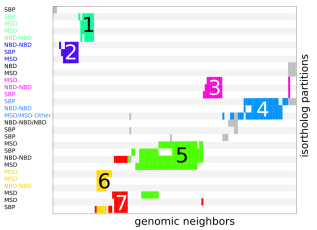
Nous allons ensuite ajouter (en colonne 6) le numéro de la CC de chaque sommet. Il nous faut maintenant croiser le gène d'ancrage qui est utilisé comme identifiant du contexte génomique du système ABC (colonne 1) avec le numéro de la CC (colonne 6). On obtient une matrice contenant 1 dans la cellule (x, y) si un gène du système x est présent dans la CC y et 0 dans tous les autres cas.
all_name <- V(graph)$name cc_ass_class <- data.frame(row.names=all_name) cc_ass_class[,1:5] <- annotation[V(graph)$name, 1:5] cc_name_list <- c() for ( i in 1:nb_cc ) { list_name <- V(cc[[i]])$name cc_name_list <- append(cc_name_list, list_name) cc_ass_class[list_name,6] <- i } color <- c('white', 'black', 'grey') matrix <- as.matrix(cc_ass_class[cc_name_list,]) mat <- table(matrix[,1], matrix[,6]) mat[mat>1] <- 1 # recouvrement entre systèmes heatmap(mat, scale='none', col=color)
* Que pouvez-vous conclure?
Décomposition en communautés
Nous allons représenter graphiquement le résultat du découpage en communautés réalisée par fastgreedy, uniquement pour le CC qui ont un taux de paralogie supérieur à 0.
* Pouvez-vous justifier ce choix ?
Pour la suite nous allons conserver les graphes modifiés pour être compatibles avec les méthodes de communautés et les dispositions pour faciliter les comparaisons entre les méthodes (utilisation de listes).
GLayout_list <- list(); # liste des layout for ( i in 1:nb_cc ) { GLayout_list[[i]] <- layout.fruchterman.reingold(cc[[i]]); }
Fastgreedy
La fonction fastgreedy.community() implémente la méthode fast greedy de Clauset, Newman et Moore. Elle retourne un objet communities de igraph qui renferme toutes les informations du partitionnement du graphe, en particulier la structure en communautés et la modularité.
Une composante connexe
Exemple pour une CC
i <- 1; com <- fastgreedy.community(cc[[i]], merges=TRUE, modularity=TRUE); Q <- modularity(com) classe_size <- length(membership(com)) method <- algorithm(com) nb_classe <- nrow(sizes(com)) title <- paste('CC ', i, ' Q=', round(Q,3), ' Size=', nb_classe, sep='') plot(com, cc[[i]], layout=GLayout_list[[i]], vertex.color= membership, vertex.label=NA, vertex.size= vertex.size, main=title) mtext(method, line=3, side=3)
Vous pouvez suivre l'évolution de la modularité en fonction des pas de l'algorithme:
plot(com$modularity)
Pour une meilleure présentation:
m <- matrix(c(1,1,2,1), 2, 2) layout(m, widths=c(2,1), heights=c(1,2)) plot(cc[[i]], layout=GLayout_list[[i]], vertex.color= membership, vertex.label=NA, vertex.size= vertex.size,main=title); mtext(method, line=3, side=3); plot(com$modularity, xlab='step', ylab='Q')
Toutes les composantes connexes
Réaliser le découpage en communautés pour toutes le CC dont le taux de paralogie est supérieur à 0.
Nous utiliserons la fonction layout() pour découper notre graphique en 6 plages.
layout(matrix(1:6,3,2)); for (i in 1:nb_cc) { if (paralogs[i,2]>0) { com <- fastgreedy.community(cc[[i]], merges=TRUE, modularity=TRUE) Q <- modularity(com) method <- algorithm(com) size <- nrow(sizes(com)) title <- paste('CC ', i, ' Q=', round(Q,3), ' Size=', size, sep='') plot(com, cc[[i]], layout=GLayout_list[[i]], vertex.color= membership, vertex.label=NA, vertex.size= vertex.size, vertex.shape=vertex.shape, main=title) } }
Représentation sous la forme de heatmap()
Annotation des communautés
# initialisations all_name <- V(graph)$name cc_ass_class <- data.frame(row.names=all_name) cc_ass_class[,1:5] <- annotation[V(graph)$name, 1:5] cc_name_list <- c() # boucle sur les CC for ( i in 1:nb_cc ) { list_name = V(cc[[i]])$name # liste des protéines présentes dans la CC cc_name_list <- append(cc_name_list, list_name) cc_ass_class[list_name,6] <- i # numéro de la CC cc_ass_class[list_name,7] <- i*100 # combiner numéro de CC avec numéro de communauté if ( paralogs[i, 2] > 0 ) { # reprendre le code pour fastgreedy cc_ass_class[list_name,7] <- cc_ass_class[list_name,7] + membership } }
Classification automatique
Nous allons réaliser une classification automatique sur les lignes et colonnes de la matrices. Nous attendons des données de type 1/0, nous allons donc appliquer une méthode de distances adaptée (disponibles dans la library(ade4)).
Par exemple,
dist_method <- 1; # jaccard
ou
dist_method <- 9; # Phi of Pearson
Nous avons aussi le choix entre différentes méthodes de classification.
clust_method <- 'ward'
Application aux lignes de la matrice :
color <- c('white', 'black', 'grey') matrix <- as.matrix(cc_ass_class[cc_name_list,]) mat <- table(matrix[,1], matrix[,7]) mat[mat>1] <- 1 # recouvrement entre systèmes df <- as.data.frame(mat[,1:ncol(mat)]) d <- dist.binary(df, method= dist_method, diag=TRUE, upper=TRUE) cd <- hclust(d, method='ward')
Faire le même type de classification sur les colonnes de la matrice.
Tracé de la carte
Etilisez heatmap() pour représenter les résultats.
heatmap(mat,Colv=as.dendrogram(ctd), Rowv=as.dendrogram(cd), , scale='none', col=color)
* Commentez le résultat obtenu.
Découpage en classes
Nous allons maintenant essayer de découper nos systèmes en classes. Pour cela, nous allons utiliser la fonction cutree().
nb_class <- 7 ass_class <- cutree(cd, nb_class) mod_mat <- mat* ass_class color <- c('white', rainbow(15)) heatmap(mod_mat,Colv=as.dendrogram(ctd), Rowv=as.dendrogram(cd), scale='none', col=color)
* Commentez le résultat obtenu.
Création d'une fonction
Nous allons créer une fonction pour faciliter le tracé des cartes.
my_display_matrix <- function(tab, dist_method, clust_method, nb_class, method) { color <- c('white', 'black', 'grey') mat <- table(tab[,1], tab[,7]) mat[mat>1] <- 1 df <- as.data.frame(mat[,1:ncol(mat)]) d <- dist.binary(df, method= dist_method, diag=TRUE, upper=TRUE) cd <- hclust(d, method=clust_method) tmat <- t(mat) dft <- as.data.frame(tmat[,1:ncol(tmat)]) dt <- dist.binary(dft,method=dist_method, diag=TRUE, upper=TRUE) cdt <- hclust(dt,method=clust_method) ass_class <- cutree(cd, nb_class) mod_mat <- mat* ass_class color <- c('white', rainbow(nb_class)) heatmap(mod_mat,Colv=as.dendrogram(cdt), Rowv=as.dendrogram(cd), scale='none', col=color, margins = c(10, 10), cexRow = 0.01, main=method) legend("topright", '',c(0:7), text.col=color) }
Utilisation:
my_display_matrix(as.matrix(cc_ass_class[cc_name_list,]), dist_method, clust_method, nb_class, method)
Walktrap
La méthode walktrap peut être utilisée de façon similaire.
com <- walktrap.community(cc[[i]])
Betweenness
Dans la nouvelle version d'igraph, la méthode betweenness s'utilise de façon similaire.
com <- edge.betweenness.community(cc[[i]])
Spinglass
La méthode spinglass nécessite une ou plusieurs boucles imbriquées selon les paramètres explorés (cette méthode est gourmandes en CPU !).
layout(matrix(c(1:2),1,2)) my_gamma <- 1 iter <- 100 # nombre d'itérations com_list <- list() # le découpage en communautés Qiter <- array(0, c(iter)) # la modularité # boucle sur les itérations for ( k in 1:iter ) { com <- spinglass.community(cc[[1]], start.temp=1, stop.temp=0.1, cool.fact=0.95, update.rule="config", gamma=my_gamma) Qiter[k] <- modularity(com) com_list[[k]] <- com } Q <- max(Qiter) step <- which.max(Qiter) membership <- membership(com_list[[step]]) size <- length(unique(membership)) title <- paste(method,' CC ', i, ' Q=', round(Q,3), ' size=', size, ' step=', step, sep='') plot(com_list[[step]], cc[[1]], layout=GLayout_list[[1]], vertex.color= membership, vertex.label=NA, vertex.size= vertex.size, main=title) hist(Qiter, nclass=5, col='lightblue', main='modularity', xlab='Q')
Comparaison des méthodes
Partitionnement en communautés
Pour comparer les performances des différentes méthodes, nous allons les lancer séquentiellement sur chaque CC. Nous reprenons le code déjà écrit dans les questions précédentes. Les résultats seront présentés graphiquement.
# Boucle sur les méthodes # conserver les résultats de chaque méthode pour chaque CC (utilisation de listes!) CC_compile_com <- list() j <- 0; # paramètres de spinglass iter <- 10 my_gamma <- 1 # boucle sur les CC # reprendre uniquement les CC qui ont de la paralogie for ( i in 1:nb_cc ) { if ( paralogs[i, 2] > 0 ) { # initialisation pour chaque CC save_com <- list() j <- j + 1 method <- 'fastgreedy' com <- fastgreedy.community(cc[[i]]) save_com[[method]] <- com method <- 'walktrap' com <- walktrap.community(cc[[i]]) save_com[[method]] <- com method <- 'betweenness' com <- edge.betweenness.community(cc[[i]]) save_com[[method]] <- com method <- 'spinglass' com_list <- list() Qiter <- array(0, c(iter)) for ( k in 1:iter ) { com <- spinglass.community(cc[[i]], start.temp=1, stop.temp=0.1, cool.fact=0.95, update.rule="config", gamma=my_gamma) Qiter[k] <- modularity(com) com_list[[k]] <- com } Q <- max(Qiter) step <- which.max(Qiter) membership <- membership(com_list[[step]]) save_com[[method]] <- com_list[[step]] # save list CC_compile_com[[j]] <- save_com } }
Représentation sous la forme de graphes
Synthèse des partitions pour chaque CC et pour chaque méthode.
Nous allons utiliser la fonction layout() pour découper notre image graphique en 4 cellules (méthodes).
# plot des graphes par(mar=c(1, 1, 1, 1)) layout(matrix(1:4, 2, 2)) vertex.size = 15 j <- 0 for ( i in 1:nb_cc ) { if ( paralogs[i, 2] > 0 ) { j <- j + 1; save_com <- CC_compile_com[[j]] nb_method <- length(save_com) names <- names(save_com) for ( k in 1: nb_method ) { method <- names[k] title <- paste('CC ', i, ' Q=', round(modularity(save_com[[method]]),3), ' taille=', size <- length(unique(membership(save_com[[method]]))), sep='') plot(save_com[[method]], cc[[i]], layout=GLayout_list[[i]], vertex.color= membership(save_com[[method]]), vertex.label=NA, vertex.size= vertex.size, vertex.shape=vertex.shape, main=title) } } }
Croisement des partitions
Un façon de comparer des partitions est de compter le nombre de fois les paires de sommets sont dans les mêmes groupes/communautés. On calcule la matrice carrée contenant en i, j le nombre de fois que le sommet i est dans le même groupe que le sommet j avec les méthodes fastgreedy, walktrap, betweenness et spinglass (les résultats des découpages en communautés des méthodes sont conservés dans matrix, calculé à l'étape précédente). On peut présenter les résultats graphiquement avec la fonction heatmap.
Représentation sous la forme heatmap()
#initialisations j <- 0 for ( i in 1:nb_cc ) { if ( paralogs[i, 2] > 0 ) { j <- j + 1; save_com <- CC_compile_com[[j]] nb_method <- length(save_com) names = names(save_com) nb_prot <- vcount(cc[[i]]) prot_names <- V(cc[[i]])$name cross <- matrix(data = 0, nrow = nb_prot, ncol = nb_prot) dimnames(cross) <- list(prot_names, prot_names) for (c1 in 1:(nb_prot-1) ) { for (c2 in (c1+1):nb_prot) { for ( m1 in 1:(nb_method-1) ) { # ils sont ensemble avec m1 if ( membership(save_com[[names[m1]]])[c1] == membership(save_com[[names[m1]]])[c2] ) { for ( m2 in (m1+1):nb_method ) { # ils sont ensemble avec m2 if ( membership(save_com[[names[m2]]])[c1] == membership(save_com[[names[m2]]])[c2] ) { cross[c1, c2] <- cross[c1, c2] + 1; cross[c2, c1] <- cross[c2, c1] + 1; } } } } } } heatmap(cross, scale='none', symm=TRUE, keep.dendro = FALSE, col=rev(gray(0:8 / 8)), main=paste('CC=', i, sep='')) } }
*interprétez les résultat obtenus.
bi-clustering
La conservation de l'ordre des gènes peut être utilisée pour valider/consolider les partitions obtenues. Les deux informations sont représentées simultanément dans une matrice dans laquelle une cellule représente un gène, une colonne un ensemble de gènes voisins sur le chromosome et une ligne un groupe de la partition. L'application d'une méthode de bi-clustering permet de combiner les deux informations et de réaliser une nouvelle partition des données. Certains gènes ne peuvent être attribués à aucune classe.
Annotations:
- 1 AI-2 importer for E. coli and S. typhimirium
- 2 Galactose importer in E. coli
- 3 Xylose transporter in E. coli
- 4 Ribose importer in E. coli