TD2 Genome Selection Plantes
From silico.biotoul.fr
(→Exercice 2 : Définition d'amorces PCR) |
(→Exercice 3 : Analyses des plantes exprimant la construction) |
||
| (45 intermediate revisions not shown) | |||
| Line 16: | Line 16: | ||
== Exercice 1 : Recherche d'ORF sur un ARNm == | == Exercice 1 : Recherche d'ORF sur un ARNm == | ||
| - | Le criblage d'une banque d'ADNc de l'oomycète a permis très probablement d'identifier la séquence nucléique correspondant à la protéine 'chitinase GH19' travaillée au cours du précédent TD. | + | Le criblage d'une banque d'ADNc de l'oomycète a permis très probablement d'identifier la séquence nucléique correspondant à la protéine 'chitinase GH19' travaillée au cours du précédent TD. Il faut maintenant de vérifier si cette séquence code effectivement pour la protéine 'chitinase-GH19' afin de pouvoir l'insérer sous contrôle de séquences régulatrices adaptées au sein d'une plante d'intérêt. |
La séquence obtenue après criblage de la banque d'ADNc est disponible ci-dessous | La séquence obtenue après criblage de la banque d'ADNc est disponible ci-dessous | ||
| Line 117: | Line 117: | ||
--> | --> | ||
| + | |||
== Exercice 2 : Définition d'amorces PCR == | == Exercice 2 : Définition d'amorces PCR == | ||
| Line 123: | Line 124: | ||
Il faut donc définir des amorces pour faire une PCR. | Il faut donc définir des amorces pour faire une PCR. | ||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | * Quel est le cahier des charges par défaut de la | + | A partir de votre séquence d'ADNc, faites une recherche d’amorces PCR avec le programme '''Primer3''' disponible [https://primer3.ut.ee/ ici]. <br> |
| - | * Obtenez vous un couple | + | <br> |
| - | * | + | * Quel est le cahier des charges par défaut de la définition d'amorces PCR avec Primer 3 ? |
| + | * Obtenez vous un couple d'amorces (primers) PCR dans la zone d'intérêt ? | ||
| + | * Pouvez-vous identifiez un couple de primers avec un Tm=55°C comme optimal (min 52, max 60) dans votre zone d'intéret ? | ||
| - | + | <br> | |
| + | Vous disposez au laboratoire du couple d'amorces ci-dessous, est-il adapté à votre expérience ? | ||
| - | + | sens : AATCAGCCTATCAACCCAAG <br/> | |
| - | + | reverse: TGTTTTGATGCATACCCACT <br/> | |
| - | sens : | + | |
| - | reverse : | + | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | <!-- | |
| + | Le couple de primers ne fonctionne qu'avec Tm=55°C comme optimal (min 52, max 60) | ||
| - | = | + | ''Paramétrez le programme pour sélectionner au mieux la zone que vous voulez amplifier (= '''la phase ouverte de lecture''') en conservant les paramètres par défaut du logiciel <br> |
| + | ''Il faudra définir la zone que vous voulez amplifier dans Targets. Le programme demande : ''position_début'', ''longueur_de_la_zone''.<br> | ||
| + | ''Exemple: Targets : 40,180 <=> on veut amplifier depuis la position 40 jusqu'à la position 220 (40+180)'''''' | ||
| + | --> | ||
| - | + | == Exercice 3 : Analyses des plantes exprimant la construction == | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | Trois plantes génétiquement modifiées et possédant la construction précédente ont été obtenues (Trans1, Trans2 et Trans3). | |
| + | Il faut maintenant évaluer si la construction 'chitinase' présente un avantage pour la plante. <br> | ||
| + | Deux expériences ont été réalisés avec avec le champignon phytopathogène ''Fusarium solani'' et le champignon opportuniste ''Candida albicans''. | ||
| - | + | Les résultats des différentes expériences sont présentés [[File:ResultsCHN2.jpg|800px|]] | |
| - | + | * Quel était l'objectif de l'expérience de la figure 1 ? | |
| + | * Interprétez la figure 1 | ||
| + | * Quel contrôle est manquant dans la Figure 1 ? | ||
| + | * Comment pouvez-vous expliquez les résultats obtenus ? | ||
| - | + | * Quelle hypothèse a conduit à mener l'expérience de la figure 2 ? | |
| + | * Interprétez la figure 2 | ||
| - | + | ** Conclure quant à l'intérêt des CHN dans des applications de biotechnologie végétale | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | * | + | |
| - | + | ||
| - | * | + | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
Current revision as of 19:25, 7 December 2022
Contents |
Objectifs
Ce TD a pour but de vous familiariser avec les méthodes de comparaison de séquences biologiques, la recherche de séquences similaires (BLAST), la recherche d'ORF dans une séquence et de mettre en application votre savoir-faire.
Quelques liens utiles:
Contexte
Nous resterons dans le contexte scientifique du précédent TD s'intéressant aux chitinases afin de définir si ces enzymes pourraient avoir un intérêt dans des approches de biotechnologie végétale. On s'intéressera ici à identifier une séquence nucléique de chitinase afin de l'introgresser dans un génome végétal sous contrôle de séquences régulatrices adaptées et d'évaluer si cette approche pourrait permettre de favoriser la protection des plantes lors d'une infection microbienne.
Exercice 1 : Recherche d'ORF sur un ARNm
Le criblage d'une banque d'ADNc de l'oomycète a permis très probablement d'identifier la séquence nucléique correspondant à la protéine 'chitinase GH19' travaillée au cours du précédent TD. Il faut maintenant de vérifier si cette séquence code effectivement pour la protéine 'chitinase-GH19' afin de pouvoir l'insérer sous contrôle de séquences régulatrices adaptées au sein d'une plante d'intérêt.
La séquence obtenue après criblage de la banque d'ADNc est disponible ci-dessous
>Oom_cDNA
ACAACATATCCAGTCACTATGGGGCCCGTCGACCTGCAGACTGGCTGTGTATAAGGGAGC CTGACATTTATATTCCCCAGAACATCAGGTTAATGGCGTTTTTGATGTCATTTTCGCGGT GGCTGAGATCAGCCACTTCTTCCCCGATAACGGAGACCGGCACACTGGCCATATCGGTGG TCATCATGCGCCAGCTTTCATCCCCGATATGCACCACCGGGTAAAGTTCACGCAAGCAAG CAGCCAAATCAGCCTATCAACCCAAGTCAGCCATGAAATTCGTCGGGGTTATCGCGTCAA GTCTTCTGGTTGTGCCTTCTGCGGTCTCTGGTGACGCTGATAGCTCGAGTTTCGCTCGCT TCTTCGATCAGGATCGTTTCCAGGAGGTTTTCCCGGACGCTGTGGAGCTCTACAACTTCA ACGGTCTTGTGGATGCGGCCAGCAAGTACAGCGAATTCGCTAATACGGGCAACGACGACA ATGACAAGCGTGAGCTGGCAGCGTTCCTGGCTCAAACAGCTCACGAGTGCGACAGCTTCA AGGCCGCGGAAGAGTACGCCCGTGACACCTACTCGGTGTGGCAGTACTGCGACAACGCCA CCTACACGTGTGCCCCCGGTCGCCGTTACCACGGCCGTGGCCCCATTCAGCTCTCATGGA ACTACAATTACTACAATGCTGGCGAAGCTCTGGGCATTGATCTCTTAAACAACCCGGACA TCGTCGCGACAGACACGACGGTGACGTGGATGACTGCGCTTTGGTACTGGATGACTCCGC ATGGCGGCCGTGTGATCCACGACATCGTCGCCGGTGAGAACGGATTCGCTCAATCCACCG ATATCATCAACGGTGGTCTGGAGTGCGGTCCGGACGCTCCCAACACGTCGAACGAGCAAC AACGTATCACGTACTTCACCAAGATGTGCGAGGCTCTGGGCGTGGAGCCTCTGGGCGCCA CCTCGTGCAACGCCTAGAGTGGGTATGCATCAAAACAAGTTTTACAAAGTAGTGAATAAG CAAAAAAGACTTTGCTTGTATTTGTGGCAGCTCCCCTTAAACGCCAGCTTTCATCCCCGA TATGCACCACCGGGTAAAGTTCACGAAGGGCGAGGAGGATAACATGGCCATCATCAAGGA GTTCATGCGCTTCAAGGTGCACATGGAGGGCTCCGTGAACGGCCACGAGTTCGAGATCGA GGGCGAGGGCGAGGGCCGCCCCTACGAGGGCACCCAGACCGCCAAGCTGAAGGTGACCAA GGGTGGCCCCCTGCCCTTCGCCTGGGACATCCTGTCCCCTCAGTTCATGTACGGCTCCAA GGCCTACGTGAAGCACCCCGCCGACATCCCCGACTACTTGAAGCTGTCCTTCCCCGAGGG CTTCAAGTGGGAGCGCGTGATGAACTTCGAGGACGGCGGCGTGGTGACCGTGACCCAGGA CTCCTCCCTGCAGGACGGCGAGTTCATCTACAAGGTGAAGCTGCGCGGCACCAACTTCCC CTCCGACGGCCCCGTAATGCAGAAGAAGACTATGGGCTGGGAGGCCTCCTCCGAGCGGAT GTACCCCGAGGACGGCGCCCTGAAGGGCGAGATCAAGCAGAGGCTGAAGCTGAAGGACGG CGGCCACTACGACGCTGAGGTCAAGACCACCTACAAGGCCAAGAAGCCCGTGCAGCTGCC CGGCGCCTACAACGTCAACATCAAGTTGGACATCACCTCCCACAACGAGGACTACACCAT CGTGGAACAGTACGAACGCGCCGAGGGCCGCCACTCCACCGGCGGCATGGACGAGCTGTA CAAGTAA
Afin d'identifier si cette séquence a une similitude avec des séquences déjà répertoriées, nous allons utiliser l'outil BLAST (Basic Local Alignement Search Tool) sur le site du NCBI (colonne de droite :Popular resources => BLAST => Nucleotide BLAST)
!! Avant de lancer votre BLAST, choisissez l'option 'somewhat similar # MEGABLAST (bas de page du BLASTn)'
- La séquence d'ADNc obtenue est -elle similaire à des séquences référencées dans les banques de données ?
- A partir de quel organisme a donc été réalisé la banque d'ADNc?
- Quel est le numéro d'accession de la séquence protéique codée par la séquence ayant une similarité avec votre ADNc ?
Maintenant que vous avez vérifié la qualité et nature de votre séquence ADNc, il est nécessaire d'identifier le cadre de lecture ouvert (ORF). Pour cela, nous allons prédire les différentes ORF possible sur votre séquence en travaillant sur les différents cadre de lecture.
Pour cela nous allons utiliser l'outil ORFfinder disponible au au NCBI. (colonne de gauche > Resources List > Classement par ordre alpahabétique)
Dans les paramètres d'ORFfinder, indiquer 300nt comme taille minimale des ORFs (=100 acide aminés).
- Interprétez le graphique obtenu
- Quelle ORF vous paraît être la plus probable ? Pourquoi ?
- Comment pouvez-vous le vérifier ?
- Utilisez dans ORF Finder, l'option BLASTp contre la banque de données 'nr (non redundant protein sequences)' et identifiez si l'ORF sélectionnée est la plus probable.
Notez la position de l'ORF (taille, codon start, codon stop, taille attendue de la protéine)
Exercice 2 : Définition d'amorces PCR
Vos analyses précédentes indiquent que votre séquence est correcte, il faut maintenant amplifier la phase ouverte de lecture (ORF) afin de l'insérer dans un vecteur sous contrôle de séquences régulatrices qui permettront son expression après transformation des plantes d'intérêt.
Il faut donc définir des amorces pour faire une PCR.
A partir de votre séquence d'ADNc, faites une recherche d’amorces PCR avec le programme Primer3 disponible ici.
- Quel est le cahier des charges par défaut de la définition d'amorces PCR avec Primer 3 ?
- Obtenez vous un couple d'amorces (primers) PCR dans la zone d'intérêt ?
- Pouvez-vous identifiez un couple de primers avec un Tm=55°C comme optimal (min 52, max 60) dans votre zone d'intéret ?
Vous disposez au laboratoire du couple d'amorces ci-dessous, est-il adapté à votre expérience ?
sens : AATCAGCCTATCAACCCAAG
reverse: TGTTTTGATGCATACCCACT
Exercice 3 : Analyses des plantes exprimant la construction
Trois plantes génétiquement modifiées et possédant la construction précédente ont été obtenues (Trans1, Trans2 et Trans3).
Il faut maintenant évaluer si la construction 'chitinase' présente un avantage pour la plante.
Deux expériences ont été réalisés avec avec le champignon phytopathogène Fusarium solani et le champignon opportuniste Candida albicans.
Les résultats des différentes expériences sont présentés 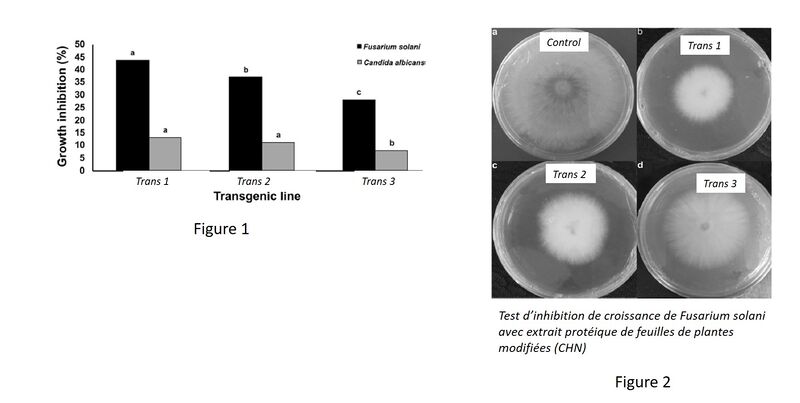
- Quel était l'objectif de l'expérience de la figure 1 ?
- Interprétez la figure 1
- Quel contrôle est manquant dans la Figure 1 ?
- Comment pouvez-vous expliquez les résultats obtenus ?
- Quelle hypothèse a conduit à mener l'expérience de la figure 2 ?
- Interprétez la figure 2
- Conclure quant à l'intérêt des CHN dans des applications de biotechnologie végétale