M1 Genomique Evolutive et Phylogenie - TP 2 Evolution proteine d'oomycete
From silico.biotoul.fr
(→Introduction) |
(→Introduction) |
||
| Line 4: | Line 4: | ||
*les algues brunes (''Ectocarpus siliculosus''). | *les algues brunes (''Ectocarpus siliculosus''). | ||
Ainsi malgré leur ressemblance morphologique, les oomycètes sont phylogénétiquement distincts des Fungi (Champignons) qui appartiennent à l’ordre des Opisthokonts. | Ainsi malgré leur ressemblance morphologique, les oomycètes sont phylogénétiquement distincts des Fungi (Champignons) qui appartiennent à l’ordre des Opisthokonts. | ||
| - | |||
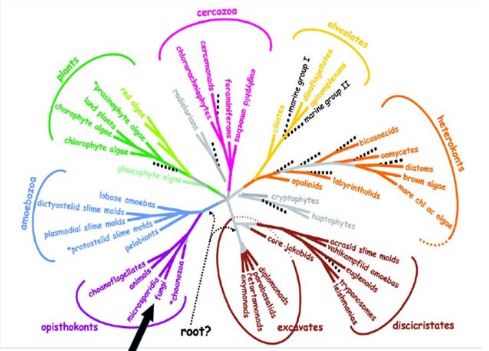
[[File:oomycete-arbre.jpg]] | [[File:oomycete-arbre.jpg]] | ||
| - | |||
| - | |||
| - | |||
Revision as of 16:49, 16 February 2017
Introduction
Les oomycètes sont des microorganismes eucaryotes filamenteux, occasionnant d’importants dégâts sur les plantes de grande culture comme le soja ou la pomme de terre. Les analyses de phylogénie moléculaire ont permis de positionner les oomycètes dans l’ordre des Heterokonts avec pour plus proches voisins
- les diatomées (Thalassossira pseudonana)
- les algues brunes (Ectocarpus siliculosus).
Ainsi malgré leur ressemblance morphologique, les oomycètes sont phylogénétiquement distincts des Fungi (Champignons) qui appartiennent à l’ordre des Opisthokonts.
De par leur position distincte par rapport aux champignons, les moyens de lutte phytosanitaire développés contre les champignons, restent peu efficaces envers les oomycètes. Ainsi nombreuses sont les recherches qui visent à mieux caractériser les acteurs moléculaires impliqués dans la virulence de ces microorganismes afin de développer des moyens de lutte plus efficace.
Objectifs et Contexte Scientifique du TP
L’équipe du Dr Jones à partir d’un milieu de culture de l’oomycète Phytophthora sojae (parasite du soja) a purifié la protéine Physo_12. L’équipe souhaite maintenant définir la fonction de cette protéine et son histoire évolutive, afin d’établir si Physo_12 pourrait être un facteur de virulence du microorganisme, c'est-à-dire une protéine qui faciliterait l’infection/colonisation de l’hôte végétal.
L’ensemble des exercices du TP a pour but de répondre à ces deux objectifs. Il est basé sur des travaux publiés par Richards et al., 2011, PNAS
Exercice 1
>Physo_12
MKVLFATALTAAAVAVASAAEFCDQWGQAKSGNYIIYNNLWGSSAANPGGKQCTALDSGSGDSVAWHTTW SWQGGDKSVKSFANAALEFDPVPLSEVKSIPSTMAYTVKSKGKAVTDVAYDLFTSSTAKGEKEFEIMIWL AALGGAGAISSTGKPIASTTIAGTEWSVYKGPNGSMMVYSFVASKQVENFEGDLLEFFNYLVKEQGFKTS QFLIKVECGTEPFVGTDVTMTVSKYSAAVNTSGGSSTPTQSGESSSSTEQTTTAPAASNTGSSAEQTTPA PAESNTGSSAEQTTPAPAGSDAGSSEEQTPSTPSTGSSTEQTTDAPAASNTGSSEETPSTGSSEETPSTG SSEETPATSDAGSSEETPATSSTGSSEETPVESSTGSSEQNPGQQEPSTPTTSSSSEETPSTETNPKCVLRRVRRE
A partir de la séquence en acide aminé de Physo_12, établissez quelle peut-être la fonction putative de cette protéine. Pour cela, suivez la procédure décrite ci-dessous
- Interrogation de banques de domaines protéiques : InterProScan
, banque de domaines
(
http://www.ebi.ac.uk/Tools/pfa/iprscan/ ) : identifiez si la proté ine Physo_12, présente des domaines répertoriés dans la banque de domaine HMM Pfam.
Relevez la
position des différents domaines. . Que si gnifie IPR002594 et PF01670
. Obtenez de l’information sur les domaines identifiés via InterProScan
. SignalP3.0
(
http://www.cbs.dtu.dk/services/SignalP/ ) : définissez si Physo_12, purifiée à partir du milieu de culture de Phytophthora sojae , est prédite
comme une protéine sécrétée.
.NCBI
(
http://www.ncbi.nlm.nih.gov/ ) : P hyso_12 est -elle déjà répertori ée dans les b anques de données. Si oui, quelle est son numéro d’ accession et son ’surnom’
dans Ge
nBank
?
. Quelles informations supplémentaires, obtenez vous sur la fiche GenPept de Physo_12
? Enregistrez au
format fasta, le transcrit de Physo_12. . Conclure